Distâncias e matrizes¶
A matriz de distância é feita através da utilização da função pdist; como já mencionado a distância euclidiana entre dois pontos \(p\) e \(q\) é \( d\left( p,q\right) = \sqrt {\sum _{i=1}^{n} \left( q_{i}-p_{i}\right)^2 }\), para \(n\) dimensões. De forma a facilitar a intuição de como esta distância é aplicada ao registo de votações vamos, passo a passo, analisar um caso simplificado (o que nos serve também de forma de validação indirecta do processo).
Vamos criar um cenário com três partidos, com nomes que reflectem o seu perfil de votação:
O Partido Favorável vota a favor.
O Partido Abstencionista abstem-se (pelo menos até certo ponto).
O Partido do Contra vota contra.
Uma votação, \( n = 1 \)¶
Começemos por uma única votação, onde cada um dos partidos segue a sua linha programática:
v1=[[1],[0],[-1]]
v1_df = pd.DataFrame(v1, columns=["v1"], index=["F","A", "C"])
v1_df
| v1 | |
|---|---|
| F | 1 |
| A | 0 |
| C | -1 |
Com uma única votação o número de dimensões é de \(n = 1\) e é bastante intuitivo que a distância entre eles é a norma:\( d\left( x,y\right) = | x - y |\); entre 1 e 0 a distância é 1, entre 1 e -1 a distância é 2, etc. Para \( n = 1 \) a distância euclidiana é equivalente à norma, pois \( \sqrt{\left( q - p\right)^2} = | q - p | \), como pode ser observado quando medidas a distância entre eles:
import math
## We could also sue the array directly, e.g.
## print("Distance from" , a[0][0] , "and" , a[1][0],"=",math.sqrt((a[0][0]-a[1][0]) ** 2))
print("Distance from", v1_df.loc["F"].name, "and", v1_df.loc["A"].name, "=",
math.sqrt((v1_df.loc["F"]["v1"]-v1_df.loc["A"]["v1"]) ** 2))
print("Distance from", v1_df.loc["F"].name, "and", v1_df.loc["C"].name, "=",
math.sqrt((v1_df.loc["F"]["v1"]-v1_df.loc["C"]["v1"]) ** 2))
print("Distance from", v1_df.loc["A"].name, "and", v1_df.loc["C"].name, "=",
math.sqrt((v1_df.loc["A"]["v1"]-v1_df.loc["C"]["v1"]) ** 2))
Distance from F and A = 1.0
Distance from F and C = 2.0
Distance from A and C = 1.0
Temos a distância entre os três possíveis pares: a distância entre F e A é idêntica à distância entre A e F. Isto é importante porque ajuda a explicar a diferença entre a forma “condensada” e forma “quadrada”. A função pdist retorna a distância entre os vários pares na forma condensada:
pdist(v1_df)
array([1., 2., 1.])
Estes são os mesmos valores que obtivemos de forma manual, e é isso que a função faz: calcula uma determinada distância (euclidiana, neste caso e por omissão) entre todos os pares possíveis, sem que seja necessário especificarmos todas as combinações possíveis.
Este formato condensado não é o que permite uma leitura mais imediata, e para tal existe a função squareform que apresenta os mesmos resultados mas numa matriz simétrica:
squareform(pdist(v1_df))
array([[0., 1., 2.],
[1., 0., 1.],
[2., 1., 0.]])
Em formato tabular torna-se ainda mais claro… e isto é exactmante a matriz de distância usada para o mapa térmico
v1_distmat=pd.DataFrame(squareform(pdist(v1)), columns=v1_df.index, index=v1_df.index)
v1_distmat
| F | A | C | |
|---|---|---|---|
| F | 0.0 | 1.0 | 2.0 |
| A | 1.0 | 0.0 | 1.0 |
| C | 2.0 | 1.0 | 0.0 |
Geometricamente temos pontos numa recta: uma análise das distâncias entre os três partidos com base no histórico de votação seria simples de visualizar com base na posição desses pontos:
fig, ax = plt.subplots()
ax.axhline(y=0,c="gold",zorder=-1)
for x in v1_df["v1"]:
ax.scatter(x,y=0,)
plt.show()

Com base na matriz de distância contruimos o mapa térmico de forma muito simples:
plt.figure(figsize=(4,4))
sns.heatmap(
v1_distmat,
cmap=sns.color_palette("Reds_r"),
linewidth=1,
annot = True,
square =True,
)
plt.show()
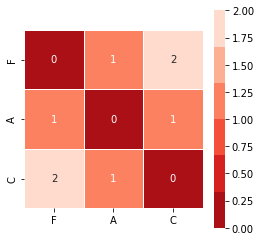
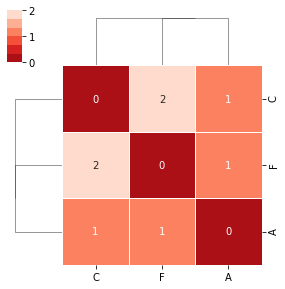
…e o agrupamento com base nessa matriz:
v1_distmat_link = hc.linkage(pdist(v1_df))
sns.clustermap(
v1_distmat,
annot = True,
cmap=sns.color_palette("Reds_r"),
linewidth=1,
row_linkage=v1_distmat_link,
col_linkage=v1_distmat_link,
figsize=(4,4)
)
plt.show()
Duas votações, \( n = 2 \)¶
Usando a mesma abordagem (agora sem necessidade de explicações adicionais) adicionamos mais uma votação, sempre em linha com o perfil fixo de votos de cada um: cada partido passa a ter dois votos, logo temos duas dimensões:
v2=[[1,1],[0,0],[-1,-1]]
v2_df = pd.DataFrame(v2, columns=["v1","v2"], index=["F","A", "C"])
v2_df
| v1 | v2 | |
|---|---|---|
| F | 1 | 1 |
| A | 0 | 0 |
| C | -1 | -1 |
A distância euclidiana é agora feita de forma mais genérica: a raíz quadrada da soma do quadrado das diferenças: para a primeira diferença isto significa, passo a passo e para \(q=F\) e \(p=A\):
\( d\left(q,p\right) = \sqrt {\sum _{i=1}^{n} \left( q_{i}-p_{i}\right)^2 } = \sqrt{\left( q_{1}-p_{1}\right)^2 + \left( q_{2}-p_{2}\right)^2 } = \sqrt{\left( 1 - 0\right)^2 + \left( 1 - 0\right)^2} = \sqrt{ 1^2 + 1^2 } = \sqrt{1+1} = \sqrt{2} \approx 1.4142135623730951 \)
E de facto:
print(math.sqrt(sum((px - qx) ** 2.0 for px, qx in zip(v2_df.loc["F"],v2_df.loc["A"]))))
print(math.sqrt(sum((px - qx) ** 2.0 for px, qx in zip(v2_df.loc["F"],v2_df.loc["C"]))))
print(math.sqrt(sum((px - qx) ** 2.0 for px, qx in zip(v2_df.loc["A"],v2_df.loc["C"]))))
1.4142135623730951
2.8284271247461903
1.4142135623730951
Tal como antes é idêntico ao resultado de pdist, tanto na sua forma condensada como quadrada:
print("pdist:\n", pdist(v2),"\n")
print("squareform:\n",squareform(pdist(v2)))
v2_distmat=pd.DataFrame(squareform(pdist(v2)), columns=v2_df.index, index=v2_df.index)
v2_distmat
pdist:
[1.41421356 2.82842712 1.41421356]
squareform:
[[0. 1.41421356 2.82842712]
[1.41421356 0. 1.41421356]
[2.82842712 1.41421356 0. ]]
| F | A | C | |
|---|---|---|---|
| F | 0.000000 | 1.414214 | 2.828427 |
| A | 1.414214 | 0.000000 | 1.414214 |
| C | 2.828427 | 1.414214 | 0.000000 |
Com duas votações temos \( n = 2 \) e conseguimos ver os pontos num espaço cartesiano em \( \mathbb{R}^2 \) (um plano).
fig, ax = plt.subplots()
for x,y in zip(v2_df["v1"], v2_df["v2"]):
ax.scatter(x,y)

A matriz de distância seria neste caso, como no anterior, desnecessária (em \( \mathbb{R}^2 \) as distâncias entre os partidos são óbvias por facilmente visualizáveis); a forma de a construir é idêntica:
plt.figure(figsize=(4,4))
sns.heatmap(
v2_distmat,
cmap=sns.color_palette("Reds_r"),
linewidth=1,
annot = True,
square =True,
)
plt.show()
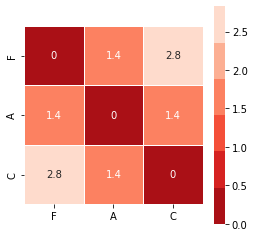
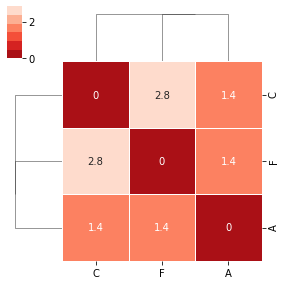
… e o clustermap que agrega distâncias e agrupamento num único gráfico:
v2_distmat_link = hc.linkage(pdist(v2_df))
sns.clustermap(
v2_distmat,
annot = True,
cmap=sns.color_palette("Reds_r"),
linewidth=1,
row_linkage=v2_distmat_link,
col_linkage=v2_distmat_link,
figsize=(4,4)
)
plt.show()
Três votações, \( n = 3 \)¶
Este é o último caso onde a visualização pode ser feita de forma directa. Consideremos:
v3=[[1,1,1],[0,0,0],[-1,-1,-1]]
v3_df = pd.DataFrame(v3, columns=["v1","v2", "v3"], index=["F","A", "C"])
v3_df
| v1 | v2 | v3 | |
|---|---|---|---|
| F | 1 | 1 | 1 |
| A | 0 | 0 | 0 |
| C | -1 | -1 | -1 |
A distância é calculada da mesma forma:
print(math.sqrt(sum((px - qx) ** 2.0 for px, qx in zip(v3_df.loc["F"],v3_df.loc["A"]))))
print(math.sqrt(sum((px - qx) ** 2.0 for px, qx in zip(v3_df.loc["F"],v3_df.loc["C"]))))
print(math.sqrt(sum((px - qx) ** 2.0 for px, qx in zip(v3_df.loc["A"],v3_df.loc["C"]))))
1.7320508075688772
3.4641016151377544
1.7320508075688772
… bem como a matriz de distância:
print("pdist:\n", pdist(v3),"\n")
print("squareform:\n",squareform(pdist(v3)))
v3_distmat=pd.DataFrame(squareform(pdist(v3)), columns=v3_df.index, index=v3_df.index)
v3_distmat
pdist:
[1.73205081 3.46410162 1.73205081]
squareform:
[[0. 1.73205081 3.46410162]
[1.73205081 0. 1.73205081]
[3.46410162 1.73205081 0. ]]
| F | A | C | |
|---|---|---|---|
| F | 0.000000 | 1.732051 | 3.464102 |
| A | 1.732051 | 0.000000 | 1.732051 |
| C | 3.464102 | 1.732051 | 0.000000 |
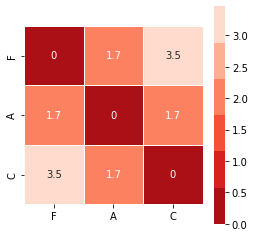
e o correspondente mapa térmico:
plt.figure(figsize=(4,4))
sns.heatmap(
v3_distmat,
cmap=sns.color_palette("Reds_r"),
linewidth=1,
annot = True,
square =True,
)
plt.show()
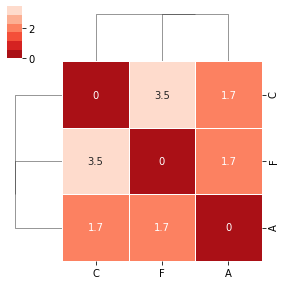
O clustermap apresenta os mesmos valores, mas reordenando colunas e linhas de forma a apresentar o resultado do agrupamento; note-se que, neste caso, não é apresentado nenhum agrupamento entre 2 dos partidos pois todos divergem de forma equidistante, constituindo três grupos separados:
v3_distmat_link = hc.linkage(pdist(v3_df))
sns.clustermap(
v3_distmat,
annot = True,
cmap=sns.color_palette("Reds_r"),
linewidth=1,
row_linkage=v3_distmat_link,
col_linkage=v3_distmat_link,
figsize=(4,4)
)
plt.show()
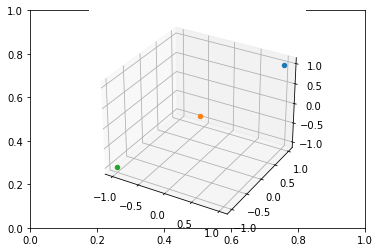
Estamos agora em \( \mathbb{R}^3 \), e para visualizar podemos usar uma projecção tridimensional:
fig, ax = plt.subplots()
ax = fig.add_subplot(111,projection='3d')
for x,y,z in zip(v3_df["v1"], v3_df["v2"], v3_df["v3"]):
ax.scatter(x,y,z)
Mais de três votações, \( n > 3 \)¶
A partir daqui coloca-se a questão fundamental: a impossibilidade de visualizar de forma directa a distância para além das três dimensões. A distância existe e segue exactamente os mesmo passos, simplesmente não é passível de visualização, razão pela qual é necessário (agora sim) depender de formas que “reduzam” as dimensões e as tornem visualizáveis.
Até agora os agrupamentos têm sido sempre iguais pois a distância é sempre linear; vamos neste último caso assumir que o Partido Abstencionista teve uma mudança de posição e passou a votar por vezes a favor e contra, embora mais contra que a favor:
v4=[[1,1,1,1,1,1,1,1,1,1],[0,0,0,1,0,-1,0,-1,-1,-1],[-1,-1,-1,-1,-1,-1,-1,-1,-1,-1]]
v4_df = pd.DataFrame(v4, columns=["v1","v2", "v3","v4","v5","v6","v7","v8","v9","v10"], index=["F","A", "C"])
v4_df
| v1 | v2 | v3 | v4 | v5 | v6 | v7 | v8 | v9 | v10 | |
|---|---|---|---|---|---|---|---|---|---|---|
| F | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 |
| A | 0 | 0 | 0 | 1 | 0 | -1 | 0 | -1 | -1 | -1 |
| C | -1 | -1 | -1 | -1 | -1 | -1 | -1 | -1 | -1 | -1 |
A distância calculdada “manualmente”:
print(math.sqrt(sum((px - qx) ** 2.0 for px, qx in zip(v4_df.loc["F"],v4_df.loc["A"]))))
print(math.sqrt(sum((px - qx) ** 2.0 for px, qx in zip(v4_df.loc["F"],v4_df.loc["C"]))))
print(math.sqrt(sum((px - qx) ** 2.0 for px, qx in zip(v4_df.loc["A"],v4_df.loc["C"]))))
4.58257569495584
6.324555320336759
3.0
… e o cálculo via pdist e a matriz de distância:
print("pdist:\n", pdist(v4),"\n")
print("squareform:\n",squareform(pdist(v4)))
v4_distmat=pd.DataFrame(squareform(pdist(v4)), columns=v4_df.index, index=v4_df.index)
v4_distmat
pdist:
[4.58257569 6.32455532 3. ]
squareform:
[[0. 4.58257569 6.32455532]
[4.58257569 0. 3. ]
[6.32455532 3. 0. ]]
| F | A | C | |
|---|---|---|---|
| F | 0.000000 | 4.582576 | 6.324555 |
| A | 4.582576 | 0.000000 | 3.000000 |
| C | 6.324555 | 3.000000 | 0.000000 |
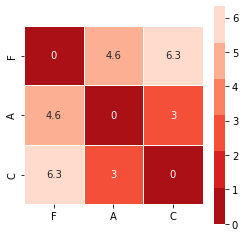
O mapa térmico:
plt.figure(figsize=(4,4))
sns.heatmap(
v4_distmat,
cmap=sns.color_palette("Reds_r"),
linewidth=1,
annot = True,
square =True,
)
plt.show()
… e o dendograma, já apresentando um agrupamento com base na maior aproximação com base no registo de votação:
v4_distmat_link = hc.linkage(pdist(v4_df), method="ward")
sns.clustermap(
v4_distmat,
annot = True,
cmap=sns.color_palette("Reds_r"),
linewidth=1,
row_linkage=v4_distmat_link,
col_linkage=v4_distmat_link,
figsize=(4,4)
)
plt.show()
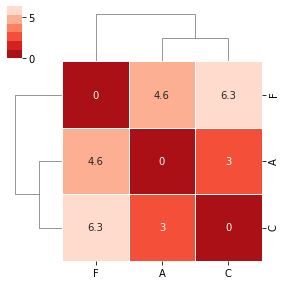
E aqui pode ser observado o resultado (e a utilidade) do cálculo das distâncias e da utilização de clustering: ao observarmos os votos acima conseguimos, intuitivamente, determinar que o partido do A está mais próximo do C, mas fazer o mesmo para 100, 1000 ou 10000 votações seria manifestamente mais difícil.
