4. Continuidade e fim: a XIV Legislatura¶
Alguns tinham ganho a partida, outros tinham perdido. Muitos tinham vindo buscar dor, outros a honra e a glória. Mas agora é tempo de os apartar - ninguém aqui consegue ver coisa nenhuma!
—Wolfram von Eschenback, «Parsival»
4.1. As eleições legislativas de 2019¶
A XIV Legislatura resultou das eleições legislativas de 2019. Estas eleições são marcadas pela subida do PS (no Governo) e perdas de praticamente todos os partidos: sem impacto no número dos deputados, no caso do BE, mas com impacto significativo no caso da CDU (que perde 10 deputados). Á direita, o CDS-PP perde 13 deputados e o PSD 10.
É uma legislatura marcada pelo crescimento de partidos recentes que já tinham representação parlamentar (o caso do PAN, que sobe de 1 para 3 deputados) e sobretudo pela estreia de dois partidos na Assembleia: a Iniciativa Liberal, o CHEGA e o Livre, ambos com 1 deputado.
4.2. Os dados das votações¶
Para a XIV legislatura os dados analisados podem incluir tanto Iniciativas como Actividades; de forma a manter uma uniformidade com os dados da XIII licenciatura irão ser usados, na análise principal, apenas os dados das Iniciativas, sendo os dados isolados ou agregados das Actividades analisados à margem.
Total de votações: 3746
Data limite inferior: 2019-10-25
Data limite superior: 2021-10-27
O processamento inicial resulta num conjunto bastante alargado de colunas e observações (votações, uma por linha), nomeadamente os votos dos vários partidos; as deputadas independentes Joacina Katar Moreira e Cristina Rodrigues têm os seus votos complementados pelos do partido de origem.
with pd.option_context("display.max_columns", 0):
display(votes[["resultado", "id"] + parties])
| resultado | id | BE | PCP | PEV | L/JKM | PS | PAN | PAN/CR | PSD | IL | CDS-PP | CH | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | Aprovado | 88184 | Contra | Contra | Contra | Contra | A Favor | Contra | Contra | A Favor | Abstenção | Contra | Contra |
| 1 | Aprovado | 88183 | Contra | Contra | Contra | Contra | A Favor | Contra | Contra | A Favor | Abstenção | Contra | Contra |
| 2 | Aprovado | 88181 | Contra | Contra | Contra | Contra | A Favor | Contra | Contra | A Favor | Abstenção | Contra | Contra |
| 3 | Aprovado | 87725 | A Favor | A Favor | A Favor | A Favor | Contra | A Favor | A Favor | A Favor | A Favor | A Favor | A Favor |
| 4 | Aprovado | 88346 | A Favor | A Favor | A Favor | A Favor | Contra | A Favor | A Favor | A Favor | A Favor | A Favor | A Favor |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 3741 | Aprovado | 92037 | A Favor | A Favor | A Favor | A Favor | Abstenção | A Favor | A Favor | A Favor | A Favor | A Favor | A Favor |
| 3742 | Aprovado | 91922 | A Favor | Ausência | NaN | NaN | A Favor | A Favor | A Favor | A Favor | NaN | A Favor | NaN |
| 3743 | Rejeitado | 92026 | Contra | Contra | Contra | Abstenção | A Favor | Abstenção | Abstenção | Contra | Contra | Contra | Contra |
| 3744 | Aprovado | 91915 | A Favor | NaN | NaN | NaN | A Favor | NaN | NaN | A Favor | NaN | A Favor | NaN |
| 3745 | Aprovado | 91916 | A Favor | NaN | NaN | NaN | A Favor | NaN | NaN | A Favor | NaN | A Favor | NaN |
3746 rows × 13 columns
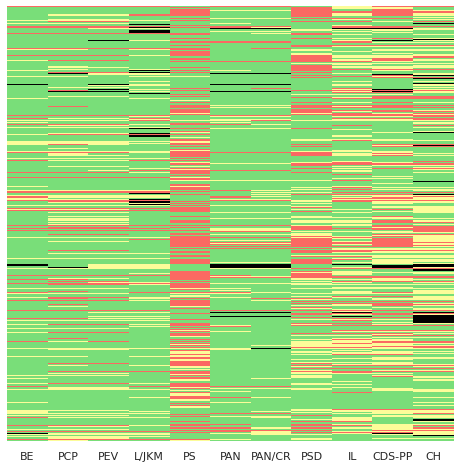
4.3. Mapa térmico das votações¶
O mapa térmico de votações para a legislatura – recordemos que nos permite ver através de cores todas as votações, dando uma imagem geral do comportamento dos vários partidos – é o seguinte:
votes_hmn = votes_hm.replace(["A Favor", "Contra", "Abstenção", "Ausência"], [1,-1,0,-2]).fillna(0)
##RYG
voting_palette = ["black","#FB6962","#FCFC99","#79DE79",]
fig = plt.figure(figsize=(8,8))
sns.heatmap(votes_hmn ,
square=False,
yticklabels = False,
cbar=False,
cmap=sns.color_palette(voting_palette),
linewidths=0,
)
plt.show()
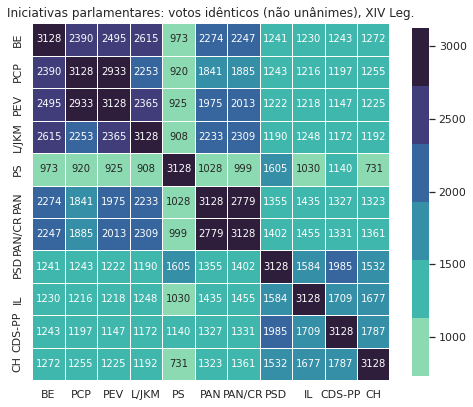
4.4. Votações idênticas¶
Das votações da legislatura é esta a matriz de votações idênticas:
pv_list = []
def highlight_diag(df):
a = np.full(df.shape, '', dtype='<U24')
np.fill_diagonal(a, 'font-weight: bold;')
return pd.DataFrame(a, index=df.index, columns=df.columns)
## Not necessarily the most straightforard way (check .crosstab or .pivot_table, possibly with pandas.melt and/or groupby)
## but follows the same approach as before in using a list of dicts
for party in votes_hm.columns:
pv_dict = collections.OrderedDict()
for column in votes_hmn:
pv_dict[column]=votes_hmn[votes_hmn[party] == votes_hmn[column]].shape[0]
pv_list.append(pv_dict)
pv = pd.DataFrame(pv_list,index=votes_hm.columns)
pv.style.apply(highlight_diag, axis=None)
| BE | PCP | PEV | L/JKM | PS | PAN | PAN/CR | PSD | IL | CDS-PP | CH | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| BE | 3746 | 2987 | 3024 | 3107 | 1582 | 2844 | 2807 | 1848 | 1762 | 1809 | 1673 |
| PCP | 2987 | 3746 | 3467 | 2749 | 1522 | 2416 | 2448 | 1843 | 1753 | 1768 | 1656 |
| PEV | 3024 | 3467 | 3746 | 2912 | 1451 | 2516 | 2542 | 1746 | 1769 | 1693 | 1658 |
| L/JKM | 3107 | 2749 | 2912 | 3746 | 1400 | 2734 | 2798 | 1680 | 1814 | 1668 | 1643 |
| PS | 1582 | 1522 | 1451 | 1400 | 3746 | 1593 | 1554 | 2221 | 1564 | 1705 | 1128 |
| PAN | 2844 | 2416 | 2516 | 2734 | 1593 | 3746 | 3385 | 1918 | 1977 | 1911 | 1747 |
| PAN/CR | 2807 | 2448 | 2542 | 2798 | 1554 | 3385 | 3746 | 1955 | 1985 | 1903 | 1773 |
| PSD | 1848 | 1843 | 1746 | 1680 | 2221 | 1918 | 1955 | 3746 | 2116 | 2548 | 1927 |
| IL | 1762 | 1753 | 1769 | 1814 | 1564 | 1977 | 1985 | 2116 | 3746 | 2241 | 2157 |
| CDS-PP | 1809 | 1768 | 1693 | 1668 | 1705 | 1911 | 1903 | 2548 | 2241 | 3746 | 2202 |
| CH | 1673 | 1656 | 1658 | 1643 | 1128 | 1747 | 1773 | 1927 | 2157 | 2202 | 3746 |
A visualização desta matriz através de um mapa térmico:
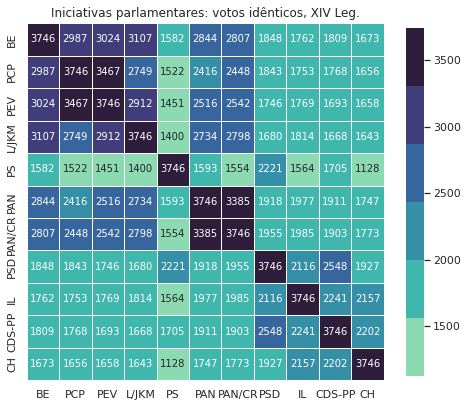
| BE | PCP | PEV | L/JKM | PS | PAN | PAN/CR | PSD | IL | CDS-PP | CH | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| BE | 3128 | 2390 | 2495 | 2615 | 973 | 2274 | 2247 | 1241 | 1230 | 1243 | 1272 |
| PCP | 2390 | 3128 | 2933 | 2253 | 920 | 1841 | 1885 | 1243 | 1216 | 1197 | 1255 |
| PEV | 2495 | 2933 | 3128 | 2365 | 925 | 1975 | 2013 | 1222 | 1218 | 1147 | 1225 |
| L/JKM | 2615 | 2253 | 2365 | 3128 | 908 | 2233 | 2309 | 1190 | 1248 | 1172 | 1192 |
| PS | 973 | 920 | 925 | 908 | 3128 | 1028 | 999 | 1605 | 1030 | 1140 | 731 |
| PAN | 2274 | 1841 | 1975 | 2233 | 1028 | 3128 | 2779 | 1355 | 1435 | 1327 | 1323 |
| PAN/CR | 2247 | 1885 | 2013 | 2309 | 999 | 2779 | 3128 | 1402 | 1455 | 1331 | 1361 |
| PSD | 1241 | 1243 | 1222 | 1190 | 1605 | 1355 | 1402 | 3128 | 1584 | 1985 | 1532 |
| IL | 1230 | 1216 | 1218 | 1248 | 1030 | 1435 | 1455 | 1584 | 3128 | 1709 | 1677 |
| CDS-PP | 1243 | 1197 | 1147 | 1172 | 1140 | 1327 | 1331 | 1985 | 1709 | 3128 | 1787 |
| CH | 1272 | 1255 | 1225 | 1192 | 731 | 1323 | 1361 | 1532 | 1677 | 1787 | 3128 |
fig = plt.figure(figsize=(8,8))
ax = fig.add_subplot()
sns.heatmap(
pv,
cmap=sns.color_palette("mako_r"),
linewidth=1,
annot = True,
square =True,
fmt="d",
cbar_kws={"shrink": 0.8})
plt.title('Iniciativas parlamentares: votos idênticos, XIV Leg.')
plt.show()
4.5. Matriz de distância e dendograma¶
Considerando a distância entre os votos (onde um voto a favor está mais perto de uma abstenção do que de um voto contra) obtemos o seguinte clustermap que conjuga a visualização da matriz de distância com o dendograma.
## Change the mapping, we now consider Abst and Aus the same
votes_hmn = votes_hm.replace(["A Favor", "Contra", "Abstenção", "Ausência"], [1,-1,0,0]).fillna(0)
## Transpose the dataframe used for the heatmap
votes_t = votes_hmn.transpose()
## Determine the Eucledian pairwise distance
## ("euclidean" is actually the default option)
pwdist = pdist(votes_t, metric='euclidean')
## Create a square dataframe with the pairwise distances: the distance matrix
distmat = pd.DataFrame(
squareform(pwdist), # pass a symmetric distance matrix
columns = votes_t.index,
index = votes_t.index
)
#show(distmat, scrollY="200px", scrollCollapse=True, paging=False)
## Normalise by scaling between 0-1, using dataframe max value to keep the symmetry.
## This is essentially a cosmetic step
#distmat=((distmat-distmat.min().min())/(distmat.max().max()-distmat.min().min()))*1
distmat.style.apply(highlight_diag, axis=None)
| BE | PCP | PEV | L/JKM | PS | PAN | PAN/CR | PSD | IL | CDS-PP | CH | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| BE | 0.000000 | 37.682887 | 33.719431 | 27.676705 | 79.956238 | 40.521599 | 40.509258 | 71.826179 | 64.202804 | 67.438861 | 59.665736 |
| PCP | 37.682887 | 0.000000 | 21.470911 | 40.224371 | 77.852424 | 52.249402 | 50.487622 | 67.712628 | 63.545259 | 65.620119 | 58.068925 |
| PEV | 33.719431 | 21.470911 | 0.000000 | 34.568772 | 78.917679 | 48.466483 | 46.690470 | 69.382995 | 63.568860 | 67.282984 | 58.557664 |
| L/JKM | 27.676705 | 40.224371 | 34.568772 | 0.000000 | 79.856121 | 40.049969 | 38.301436 | 71.407283 | 62.032250 | 66.843100 | 58.343809 |
| PS | 79.956238 | 77.852424 | 78.917679 | 79.856121 | 0.000000 | 78.885994 | 78.587531 | 58.770741 | 68.636725 | 66.294796 | 72.173402 |
| PAN | 40.521599 | 52.249402 | 48.466483 | 40.049969 | 78.885994 | 0.000000 | 22.956481 | 68.607580 | 58.086143 | 63.545259 | 56.639209 |
| PAN/CR | 40.509258 | 50.487622 | 46.690470 | 38.301436 | 78.587531 | 22.956481 | 0.000000 | 66.813172 | 56.982453 | 63.047601 | 55.506756 |
| PSD | 71.826179 | 67.712628 | 69.382995 | 71.407283 | 58.770741 | 68.607580 | 66.813172 | 0.000000 | 54.616847 | 45.000000 | 53.075418 |
| IL | 64.202804 | 63.545259 | 63.568860 | 62.032250 | 68.636725 | 58.086143 | 56.982453 | 54.616847 | 0.000000 | 47.370877 | 47.010637 |
| CDS-PP | 67.438861 | 65.620119 | 67.282984 | 66.843100 | 66.294796 | 63.545259 | 63.047601 | 45.000000 | 47.370877 | 0.000000 | 43.497126 |
| CH | 59.665736 | 58.068925 | 58.557664 | 58.343809 | 72.173402 | 56.639209 | 55.506756 | 53.075418 | 47.010637 | 43.497126 | 0.000000 |
O clustermap inclui os resultados do agrupamentos pelo método de Ward, com base nas distâncias acima:
## Perform hierarchical linkage on the distance matrix using Ward's method.
distmat_link = hc.linkage(pwdist, method="ward", optimal_ordering=True )
sns.clustermap(
distmat,
annot = True,
cmap=sns.color_palette("Reds_r"),
linewidth=1,
#standard_scale=1,
row_linkage=distmat_link,
col_linkage=distmat_link,
figsize=(8,8)).fig.suptitle('Portuguese Parliament 14th Legislature, Clustermap',y=1)
plt.show()
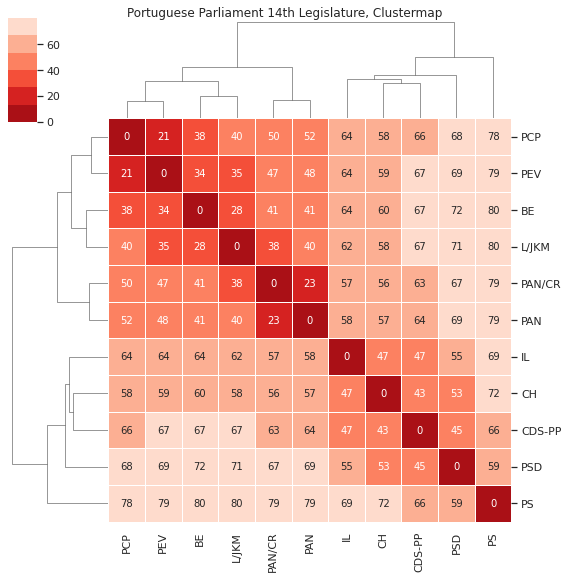
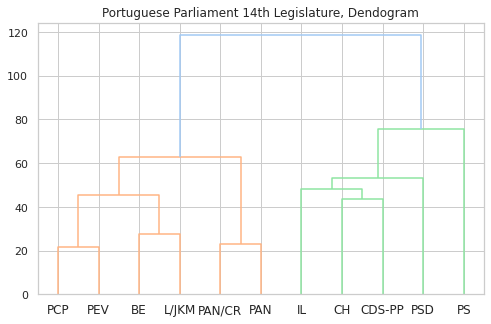
O agrupamento, também derivado do número de partidos, tem diferenças óbvias em relação ao da XIII legislatura: por um lado o posicionamento do PS, por outro a forma como os novos partidos se agrupam (entre si, e com os que já tinham representam).
4.6. Clustering de observações: DBSCAN e Spectrum Scaling¶
Uma forma diferente de determinar agrupamentos é através de métodos de clustering, que procuram determinar agrupamentos de pontos com base em mecanismos específicos de cada um dos algoritmos.
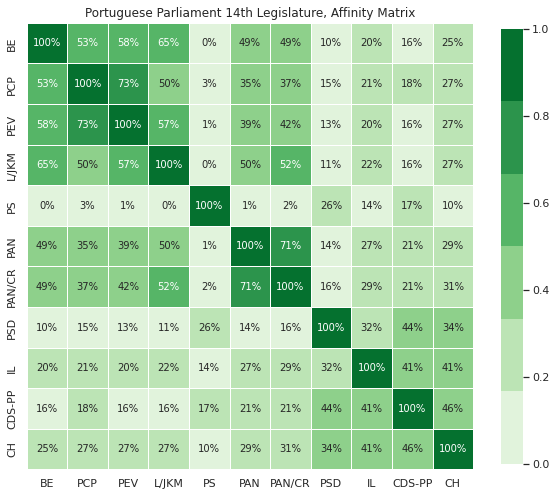
Vamos demonstrar dois deles, e como passo preliminar vamos transformar a nossa matriz de distâncias: ao contrário do dendograma anterior estes métodos utilizam uma matriz de afinidade, onde valores mais altos significam uma maior semelhança (e, consequentemente, para uma matriz simétrica a diagonal passa de 0 para 1).
Como passo preliminar normalizamos as distâncias no intervalo [0,1], após o qual obtemos a matriz de afinidade a partir da matriz de distância:
distmat_mm=((distmat-distmat.min().min())/(distmat.max().max()-distmat.min().min()))*1
#pd.DataFrame(distmat_mm, distmat.index, distmat.columns)
affinmat_mm = pd.DataFrame(1-distmat_mm, distmat.index, distmat.columns)
affinmat_mm.style.apply(highlight_diag, axis=None)
| BE | PCP | PEV | L/JKM | PS | PAN | PAN/CR | PSD | IL | CDS-PP | CH | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| BE | 1.000000 | 0.528706 | 0.578276 | 0.653852 | 0.000000 | 0.493203 | 0.493357 | 0.101681 | 0.197026 | 0.156553 | 0.253770 |
| PCP | 0.528706 | 1.000000 | 0.731467 | 0.496920 | 0.026312 | 0.346525 | 0.368559 | 0.153129 | 0.205250 | 0.179300 | 0.273741 |
| PEV | 0.578276 | 0.731467 | 1.000000 | 0.567654 | 0.012989 | 0.393837 | 0.416050 | 0.132238 | 0.204954 | 0.158502 | 0.267629 |
| L/JKM | 0.653852 | 0.496920 | 0.567654 | 1.000000 | 0.001252 | 0.499101 | 0.520970 | 0.106920 | 0.224172 | 0.164004 | 0.270303 |
| PS | 0.000000 | 0.026312 | 0.012989 | 0.001252 | 1.000000 | 0.013385 | 0.017118 | 0.264964 | 0.141571 | 0.170861 | 0.097339 |
| PAN | 0.493203 | 0.346525 | 0.393837 | 0.499101 | 0.013385 | 1.000000 | 0.712887 | 0.141936 | 0.273526 | 0.205250 | 0.291622 |
| PAN/CR | 0.493357 | 0.368559 | 0.416050 | 0.520970 | 0.017118 | 0.712887 | 1.000000 | 0.164378 | 0.287329 | 0.211474 | 0.305786 |
| PSD | 0.101681 | 0.153129 | 0.132238 | 0.106920 | 0.264964 | 0.141936 | 0.164378 | 1.000000 | 0.316916 | 0.437192 | 0.336194 |
| IL | 0.197026 | 0.205250 | 0.204954 | 0.224172 | 0.141571 | 0.273526 | 0.287329 | 0.316916 | 1.000000 | 0.407540 | 0.412045 |
| CDS-PP | 0.156553 | 0.179300 | 0.158502 | 0.164004 | 0.170861 | 0.205250 | 0.211474 | 0.437192 | 0.407540 | 1.000000 | 0.455988 |
| CH | 0.253770 | 0.273741 | 0.267629 | 0.270303 | 0.097339 | 0.291622 | 0.305786 | 0.336194 | 0.412045 | 0.455988 | 1.000000 |
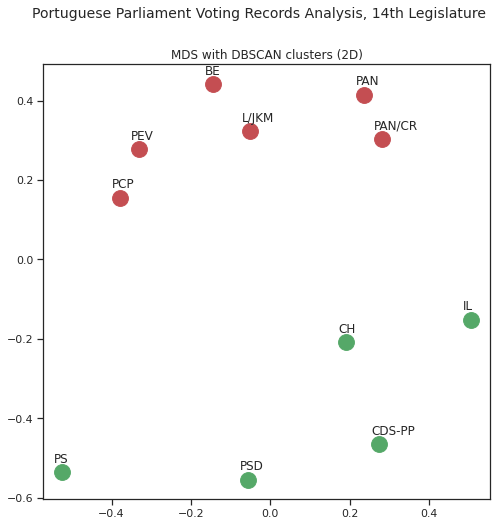
4.6.1. DBSCAN¶
O DBSCAN é algoritmo que, entre outras características, não necessita de ser inicializado com um número pré-determinado de grupos, procedendo à sua identificação através da densidade dos pontos [DBS]: o DBSCAN identifica os grupos de forma automática.
from sklearn.cluster import DBSCAN
dbscan_labels = DBSCAN(eps=0.8).fit(affinmat_mm)
dbscan_labels.labels_
dbscan_dict = dict(zip(distmat_mm,dbscan_labels.labels_))
pd.DataFrame.from_dict(dbscan_dict, orient='index', columns=["Group"]).T
| BE | PCP | PEV | L/JKM | PS | PAN | PAN/CR | PSD | IL | CDS-PP | CH | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Group | 0 | 0 | 0 | 0 | -1 | 0 | 0 | -1 | -1 | -1 | -1 |
Com DBSCAN identificamos 2 grupos, diferentes dos obtidos para a anterior legislatura.
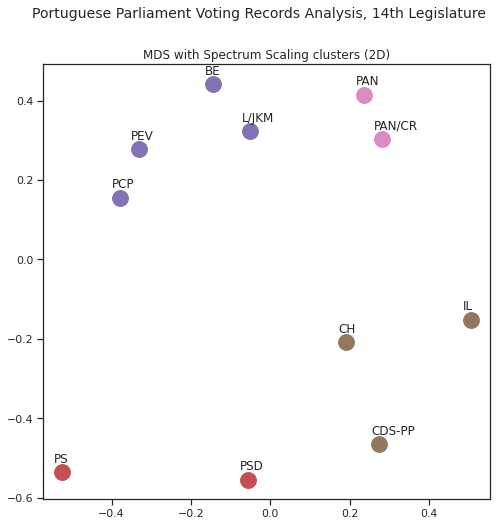
4.6.2. Spectral clustering¶
Outra abordagem para efectuar a identificação de grupos passa pela utilização de Spectral Clustering, uma forma de clustering que utiliza os valores-próprios e vectores-próprios de matrizes como forma de determinação dos grupos. Este método necessita que seja determinado a priori o número de clusters; assim, podemos usar este método para agrupamentos mais finos, neste caso identificando 4 grupos (dado o maior número de partidos e deputadas não-inscritas):
from sklearn.cluster import SpectralClustering
sc = SpectralClustering(4, affinity="precomputed",random_state=2020).fit_predict(affinmat_mm)
sc_dict = dict(zip(distmat,sc))
pd.DataFrame.from_dict(sc_dict, orient='index', columns=["Group"]).T
| BE | PCP | PEV | L/JKM | PS | PAN | PAN/CR | PSD | IL | CDS-PP | CH | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Group | 1 | 1 | 1 | 1 | 0 | 3 | 3 | 0 | 2 | 2 | 2 |
Neste caso, e por determinarmos que devem existir 4 grupos, este algoritmo agrupa PS e PSD, PEV/BE/PCP/JKM, o PAN e Cristina Rodrigues, e o CHEGA, IL e CDS-PP.
4.6.3. Multidimensional scaling¶
Não temos ainda uma forma de visualizar a distância relativa de cada partido em relação aos outros com base nas distâncias/semelhanças: temos algo próximo com base no dendograma mas existem outras formas de visualização interessantes.
Uma das formas é o multidimensional scaling que permite visualizar a distância ao projectar em 2 ou 3 dimensões (também conhecidas como dimensões visualizavies) conjuntos multidimensionais, mantendo a distância relativa [ZWH15].
from sklearn.manifold import MDS
## Graphic options
sns.set()
sns.set_style("whitegrid")
fig, ax = plt.subplots(figsize=(8,8))
plt.title('Portuguese Parliament Voting Records Analysis, 14th Legislature', fontsize=14)
for label, x, y in zip(distmat_mm.columns, coords[:, 0], coords[:, 1]):
ax.scatter(x, y, s=250)
ax.axis('equal')
ax.annotate(label,xy = (x-0.02, y+0.025))
plt.show()
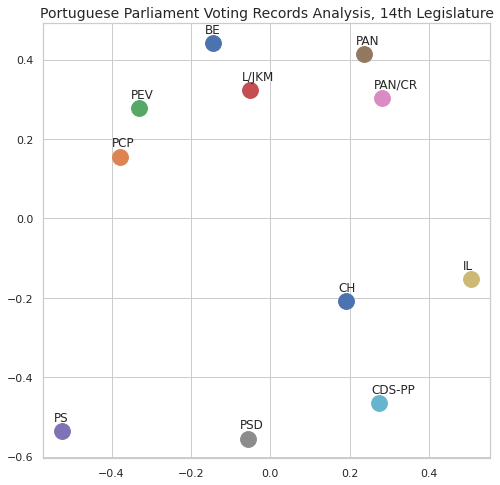
Por último, o mesmo MDS em 3D, e em forma interactiva:
mds = MDS(n_components=3, dissimilarity='precomputed',random_state=1234, n_init=100, max_iter=1000)
results = mds.fit(distmat.values)
parties = distmat.columns
coords = results.embedding_
import plotly.graph_objects as go
# Create figure
fig = go.Figure()
# Loop df columns and plot columns to the figure
for label, x, y, z in zip(parties, coords[:, 0], coords[:, 1], coords[:, 2]):
fig.add_trace(go.Scatter3d(x=[x], y=[y], z=[z],
text=label,
textposition="top center",
mode='markers+text', # 'lines' or 'markers'
name=label))
fig.update_layout(
width = 1000,
height = 1000,
title = "14th Legislature: 3D MDS",
template="plotly_white",
showlegend=False
)
fig.update_yaxes(
scaleanchor = "x",
scaleratio = 1,
)
plot(fig, filename = 'l14-3d-mds.html')
display(HTML('l14-3d-mds.html'))

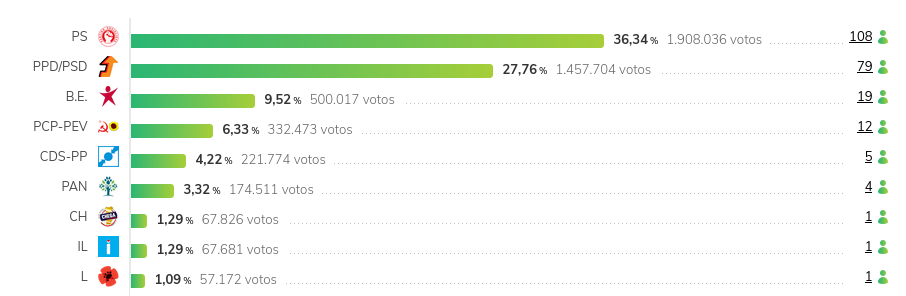 Fonte: CNE
Fonte: CNE